Dihydrolipoamide S-acétyltransférase
| Dihydrolipoamide S-acétyltransférase | ||
 Pyruvate déshydrogénase kinase 3 liée au domaine lipoyle 2 d'un complexe pyruvate déshydrogénase humain (PDB 1Y8N[1]) | ||
| Caractéristiques générales | ||
|---|---|---|
| Symbole | DLAT | |
| N° EC | 2.3.1.12 | |
| Homo sapiens | ||
| Locus | 11q23.1 | |
| Masse moléculaire | 68 997 Da[2] | |
| Nombre de résidus | 647 acides aminés[2] | |
| Liens accessibles depuis GeneCards et HUGO. | ||
La dihydrolipoamide S-acétyltransférase (E2), ou dihydrolipoyl transacétylase, est la deuxième des trois enzymes du complexe pyruvate déshydrogénase (PDC), constitué d'une décarboxylase, d'une transacétylase et d'une réductase agissant séquentiellement dans la décarboxylation oxydative du pyruvate en acétyl-CoA, réaction qui réalise notamment la liaison entre la glycolyse et le cycle de Krebs ; les autres enzymes du complexe sont la pyruvate déshydrogénase (E1) et la dihydrolipoyl déshydrogénase (E3).
Le complexe pyruvate déshydrogénase
S-acétyltransférase (E2)
| N° EC | EC 2.3.1.12 |
|---|---|
| N° CAS | 9032-29-5 |
| IUBMB | Entrée IUBMB |
|---|---|
| IntEnz | Vue IntEnz |
| BRENDA | Entrée BRENDA |
| KEGG | Entrée KEGG |
| MetaCyc | Voie métabolique |
| PRIAM | Profil |
| PDB | RCSB PDB PDBe PDBj PDBsum |
| GO | AmiGO / EGO |
modifier 
La réaction globale catalysée par le PDC est la suivante :
 | + CoA-SH + NAD+ → NADH + H+ + CO2 + |  |
| Pyruvate | Acétyl-CoA |
Le mécanisme de cette réaction est assez complexe, et peut être résumé par le schéma simplifié ci-dessous :

Mécanisme réactionnel du complexe pyruvate déshydrogénase (R = H sur ce schéma) :
- la pyruvate déshydrogénase (E1) catalyse les étapes A et B avec la thiamine pyrophosphate (TPP),
- la dihydrolipoamide S-acétyltransférase (E2) catalyse l'étape C avec le lipoamide et la coenzyme A (CoA-SH),
- la dihydrolipoyl déshydrogénase (E3) catalyse les étapes D et E avec la FAD et la NAD+.
La dihydrolipoamide S-acétyltransférase
La dihydrolipoamide S-acétyltransférase est une acétyltransférase faisant intervenir successivement trois cofacteurs : la thiamine pyrophosphate (TPP, liée à la pyruvate déshydrogénase), le dihydrolipoamide et la coenzyme A (CoA-SH), cette dernière étant convertie en acétyl-coenzyme A (acétyl-CoA).
 |  |  | ||
| Thiamine pyrophosphate (TPP) | Dihydrolipoamide | Coenzyme A (CoA-SH) |
Le véritable cofacteur est en fait l'acide lipoïque davantage que le (dihydro)lipoamide, mais cet acide carboxylique est lié par covalence à la protéine E2 au moyen d'une liaison amide sur un résidu lysine, ce qui donne l'équivalent d'un résidu lipoamide.
 | 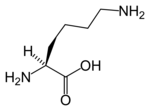 | |
| Acide lipoïque | L-Lysine |
Nomenclature et dénominations
Le nom systématique de cette enzyme est acétyl-CoA:enzyme-N6-(dihydrolipoyl)lysine S-acétyltransférase.
Dans la littérature, elle peut cependant être désignée par tout une série de noms d'usage, tels que (liste non limitative) :
- acétyl-CoA:dihydrolipoamide S-acétyltransférase,
- acide lipoïque acétyltransférase,
- dihydrolipoamide S-acétyltransférase,
- dihydrolipoate acétyltransférase,
- dihydrolipoïque transacétylase,
- dihydrolipoyl acétyltransférase,
- lipoate acétyltransférase,
- lipoate transacétylase,
- lipoïque acétyltransférase,
- lipoic transacétylase,
- lipoylacétyltransférase,
- thioltransacétylase A,
- transacétylase X.
Notes et références
- ↑ (en) Masato Kato, Jacinta L. Chuang, Shih-Chia Tso, R. Max Wynn et David T. Chuang, « Crystal structure of pyruvate dehydrogenase kinase 3 bound to lipoyl domain 2 of human pyruvate dehydrogenase complex », The EMBO Journal, vol. 24, no 10, , p. 1763-1774 (PMID 15861126, PMCID 1142596, DOI 10.1038/sj.emboj.7600663, lire en ligne)
- ↑ a et b Les valeurs de la masse et du nombre de résidus indiquées ici sont celles du précurseur protéique issu de la traduction du gène, avant modifications post-traductionnelles, et peuvent différer significativement des valeurs correspondantes pour la protéine fonctionnelle.
Articles connexes
- Dihydrolipoamide S-succinyltransférase, enzyme E2 du complexe α-cétoglutarate déshydrogénase
- Dihydrolipoyl transacylase, enzyme E2 du complexe 3-méthyl-2-oxobutanoate déshydrogénase
v · m | |
|---|---|
| 2.3.1 : groupes autres que aminoacyle (principalement acétyltransférases) |
|
| 2.3.2 : aminoacyltransférases |
|
| 2.3.3 : groupe converti en alkyle pendant le transfert |
|
 Portail de la biochimie
Portail de la biochimie












